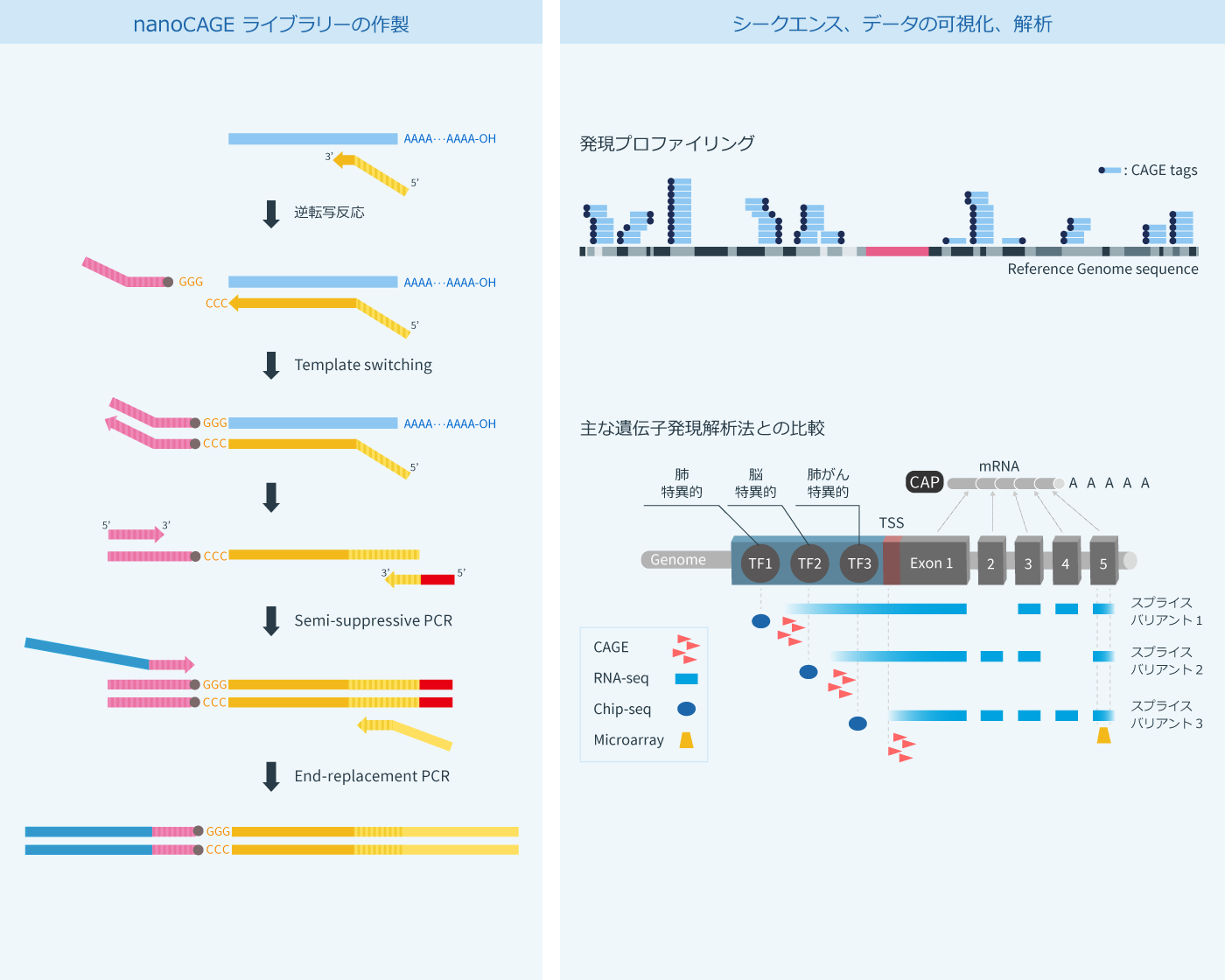
nanoCAGEライブラリー
nanoCAGE/CAGEscanライブラリー受託合成は、お客様からお預かりするtotal RNAサンプルからnanoCAGE/CAGEscanライブラリーを作製するサービスです。 従来のCAGE(Cap Analysis of Gene Expression)法では、対応できなかった微量サンプルからライブラリーを作製し、転写開始点の同定と遺伝子発現の解析を大規模に解析することが可能になりました。ダナフォームでは、シークエンス解析やデータ解析のオプションも合わせて総合的に研究開発をバックアップします。
nanoCAGE/CAGEscan法は、理化学研究所と共同開発したCAGE-seqを改良し、微量サンプルからのライブラリー作製を可能にした遺伝子発現解析法です。 nanoCAGE/CAGEscan法の際立った特徴は、CAGE-seqと同様に、転写物のプロモーターを正確にとらえて、これにもとづく発現プロファイルを取得できることです。
nanoCAGE/CAGEscan法は、RNAの5’末端から切り出した短いタグの配列を決定し、マップすることにより、タグ配列の頻度を数値化するものです。ダナフォームでは、次世代シーケンサーを用いた「CAGE-seqサービス」をご提供します。Illumina HiSeq2000/2500シーケンサーの1レーンを用いたシーケンス解析により、8-plex時に、サンプルあたり平均300万リードの解析結果が得られます。 CAGE-seqで得られた発現プロファイルは、各種発現解析やゲノムアノテーション研究における強力なツールとなります。
nanoCAGE法は解析のターゲットとなるRNA量が微量であっても遺伝子発現解析ができるため、神経系の細胞の解析や、がん細胞の解析にも適用できます。
nanoCAGE/CAGEscan法の主なアプリケーション
- 少量サンプルのゲノムワイドな遺伝子発現解析
- プロモーター部位の予測
- 半定量的mRNA/ncRNA発現プロファイル
nanoCAGE/CAGEscan法の特徴
少量サンプルに対応
必要なtotal RNAはナノグラム単位。
新たなデータ
3'側も同時に解析が可能。
ユニークなデータ
転写開始点を実験的に同定、プロモーター活性の数値化が可能。
広い応用範囲
どの生物種でもトランスクリプトーム解析が可能。
広いダイナミックレンジ
まれな転写物の検出も可能。
関連情報:
CAGE-seqとnanoCAGE/CAGEscan法の比較
| 方法 | 利点 | 欠点 |
| CAGE-seq | PCRによるバイアスのない正確な発現解析が可能。 | 解析に必要なtotal RNA:3 μg (植物などでは5μgを推奨) |
|---|---|---|
| nanoCAGE/CAGEscan | 解析に必要なtotal RNA:50 ng(推奨100ng)。 3'側も同時に解析が可能。 |
ライブラリー作製の行程でPCRのステップが含まれる。 |